基因组、cDNA、氨基酸坐标转换神器:Transvar
简介
Transvar 是 MD Anderson 开发的一款多种方向的突变/坐标转换工具,它支持基因组坐标、cDNA 坐标以及蛋白氨基酸坐标之间的转换。Google 了一下,发现对于这个工具的介绍还是很少的,于是来介绍一下。
举个实际例子,肺癌中的EGFR基因突变,在 COSMIC 上见到的一个突变的描述网页如下,我们可以看到id 为COSM6223的突变,是一个 EGFR 基因上的缺失突变,对应的 cDNA 上的突变为c.2235_2249del15,而氨基酸突变为p.E746_A750delELREA,基因组坐标则为7:55242465..55242479;那么这个突变是否属于 EGFR 19号外显子缺失呢?缺失的序列是哪些?

网页版使用
首先我们打开 Transvar 的网页版:https://bioinformatics.mdanderson.org/transvar/
基因组正向注释
首先我们来尝试从基因组正向注释到 cDNA 及氨基酸坐标上。勾选Forward Annotation: gDNA,我们可以看见示例输入如下。
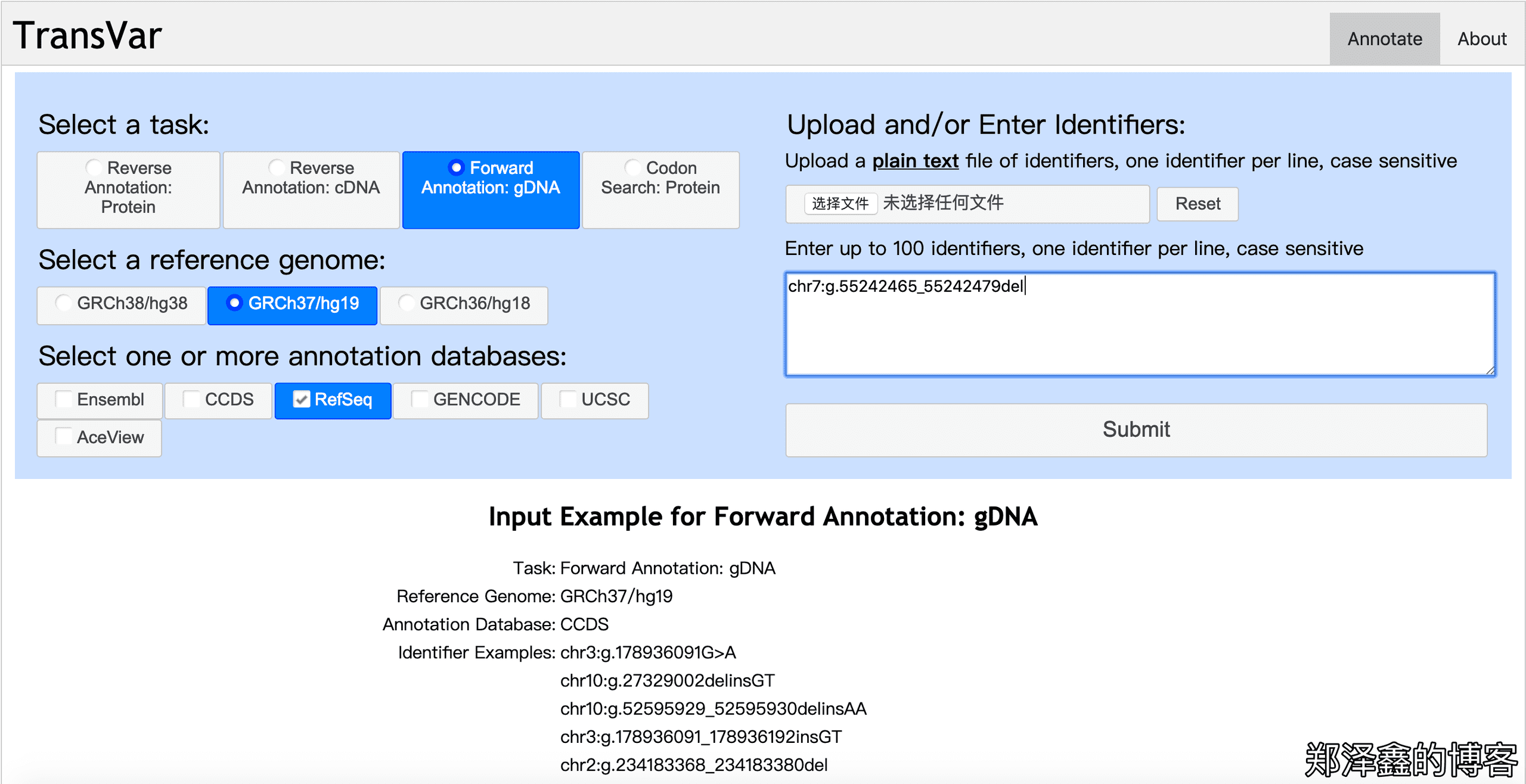
于是如上图,我们勾选GRCh37/hg19,并且勾选下面的RefSeq(需要其他数据库的可以都勾选上),在右侧的输入框中输入chr7:g.55242465_55242479del,点击Submit提交。
然后我们就能看到结果如下,
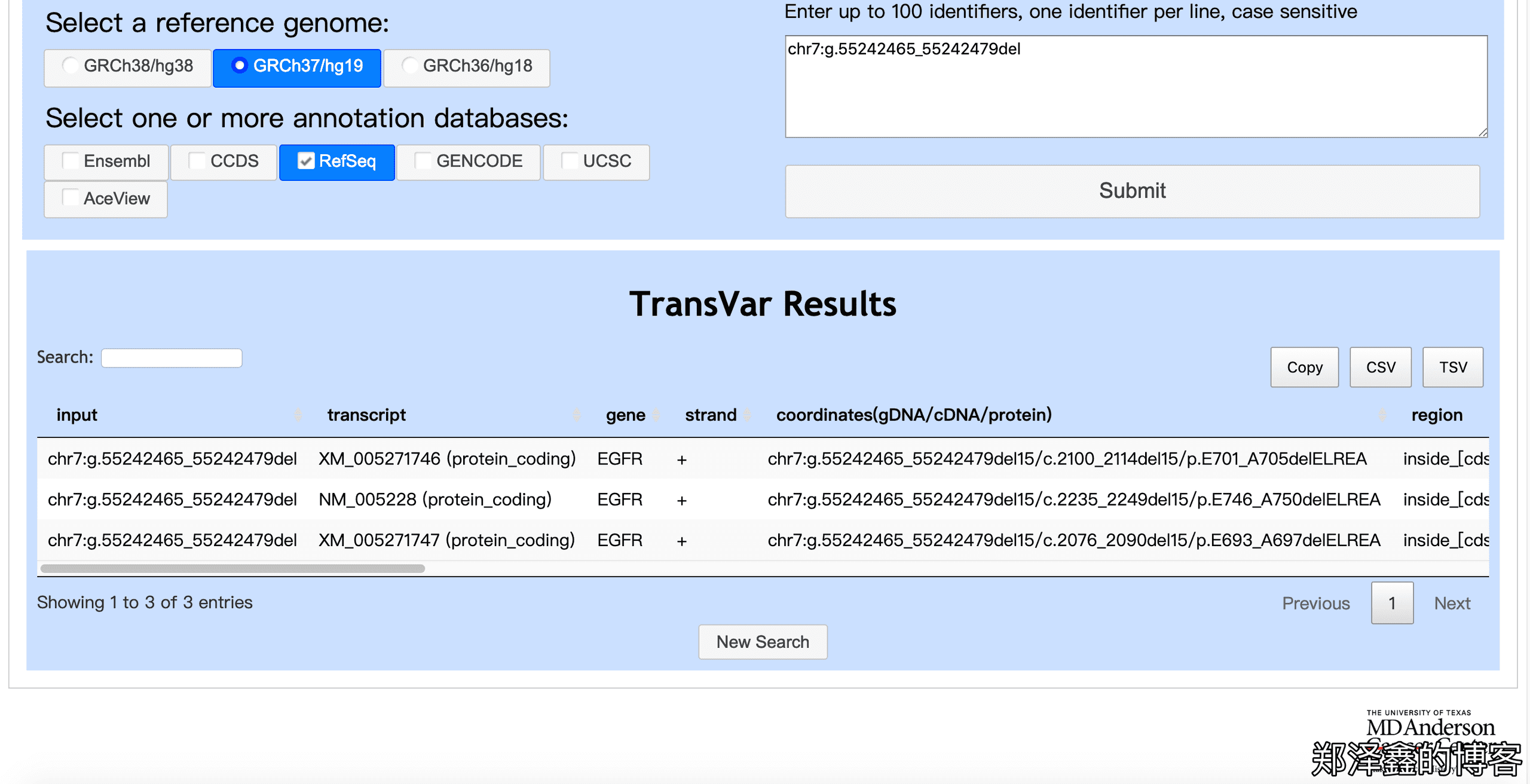
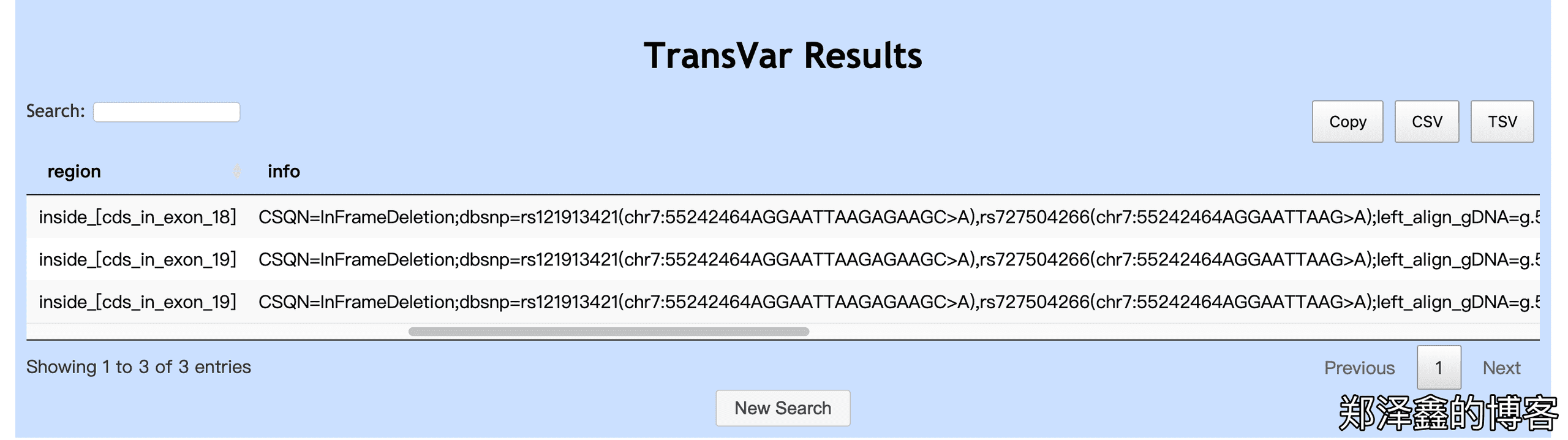
如下图,XM_开头的为预测转录本,我们只看NM_005228的结果,该突变确实为 EGFR 19号外显子缺失,并且缺失的序列为AGGAATTAAGAGAAGC>A。滑动滚动条,我们还能看到更多的注释结果。
cDNA 反向注释
尝试了基因组正向的注释,我们来测试一下通过 cDNA 坐标反向注释回基因组以及氨基酸坐标,这在我们只知道某种转录本的特定突变,需要查找基因、基因组坐标时特别有用。
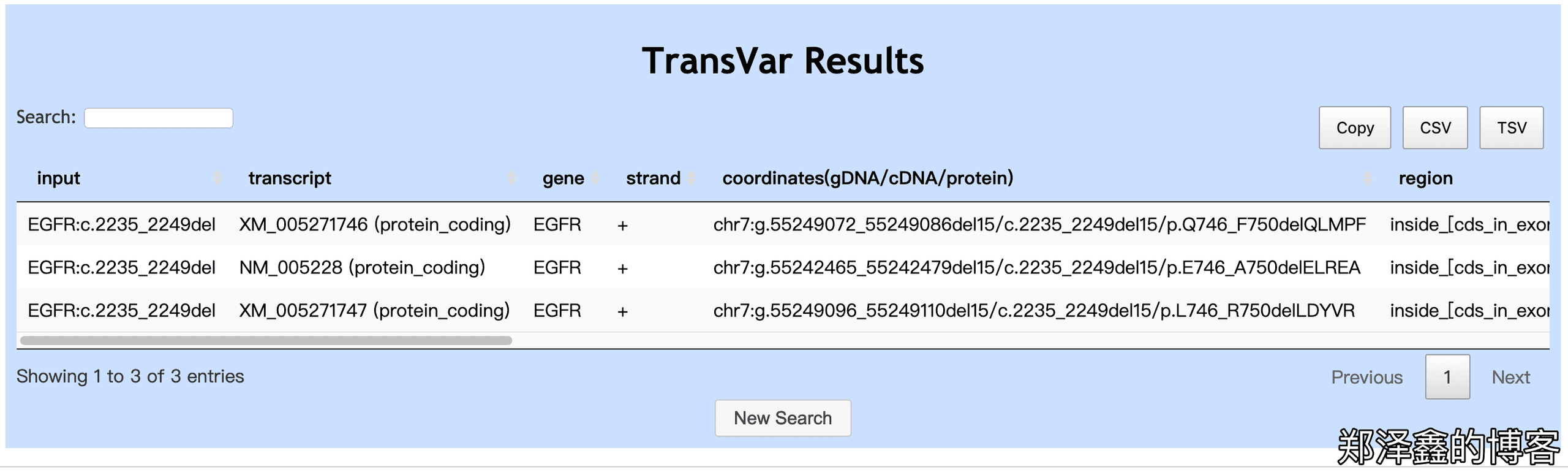
如下图,勾选Reverse Annotation: cDNA,保持勾选GRCh37/hg19以及下面的RefSeq,根据示例提示,输入EGFR:c.2235_2249del,Submit。

如下图我们可以看到结果与之前的基因组正向注释的输入、结果都是一致的。
氨基酸反向注释
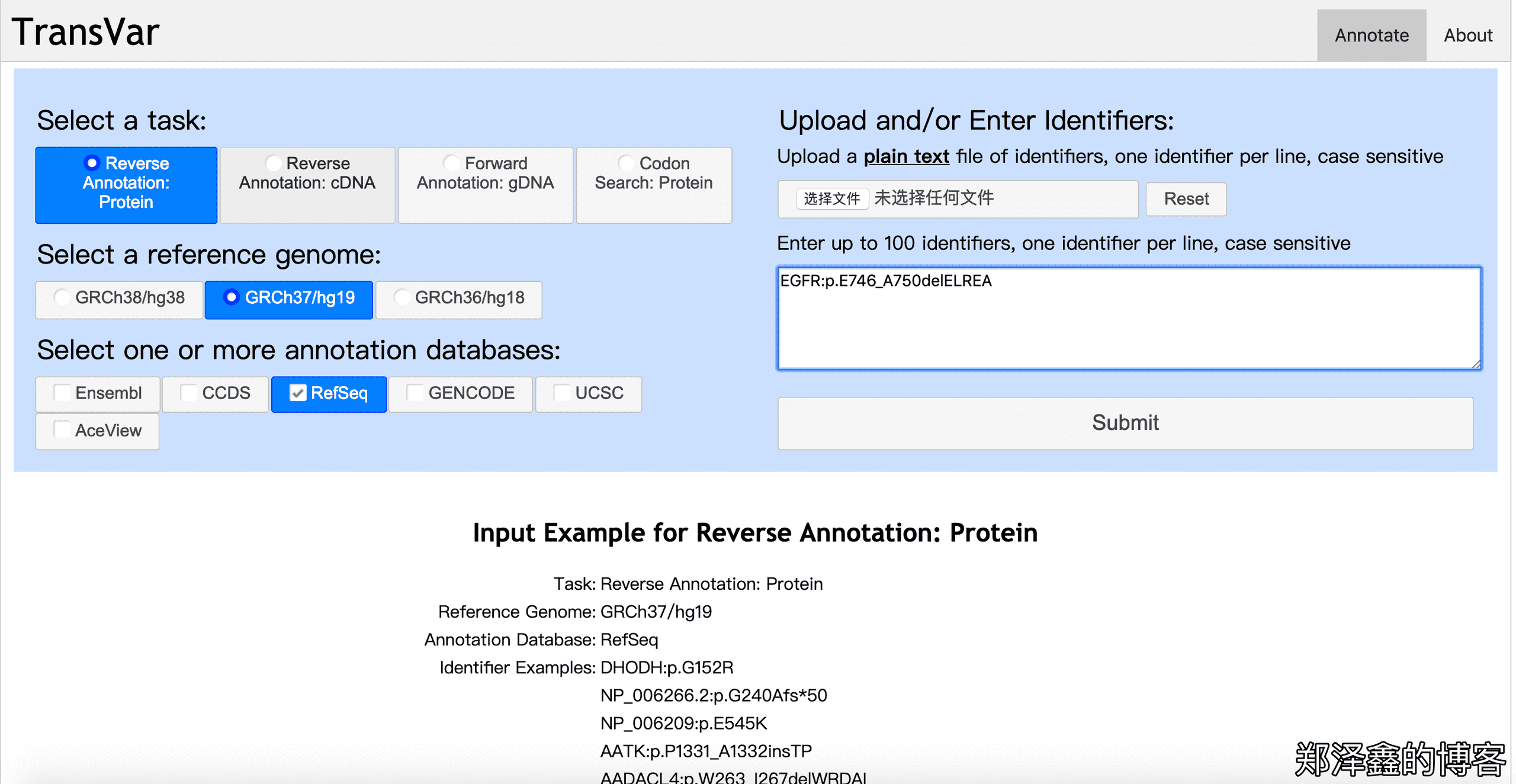
最后我们来尝试一下通过氨基酸突变的反向注释,当我们获得以氨基酸水平的突变表示时,我们可以通过 Transvar,轻松地转换成基因组/cDNA 水平的突变。如下图勾选Reverse Annotation: Protein,同样保持勾选GRCh37/hg19以及下面的RefSeq,根据示例提示,输入EGFR:p.E746_A750delELREA,Submit。
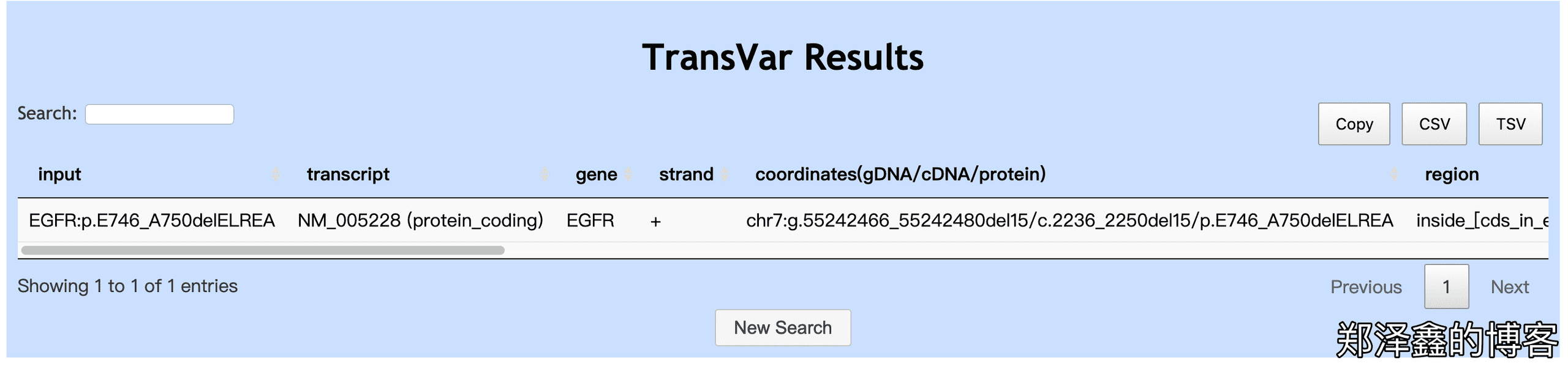
如下图,结果毫无疑问是一致的。
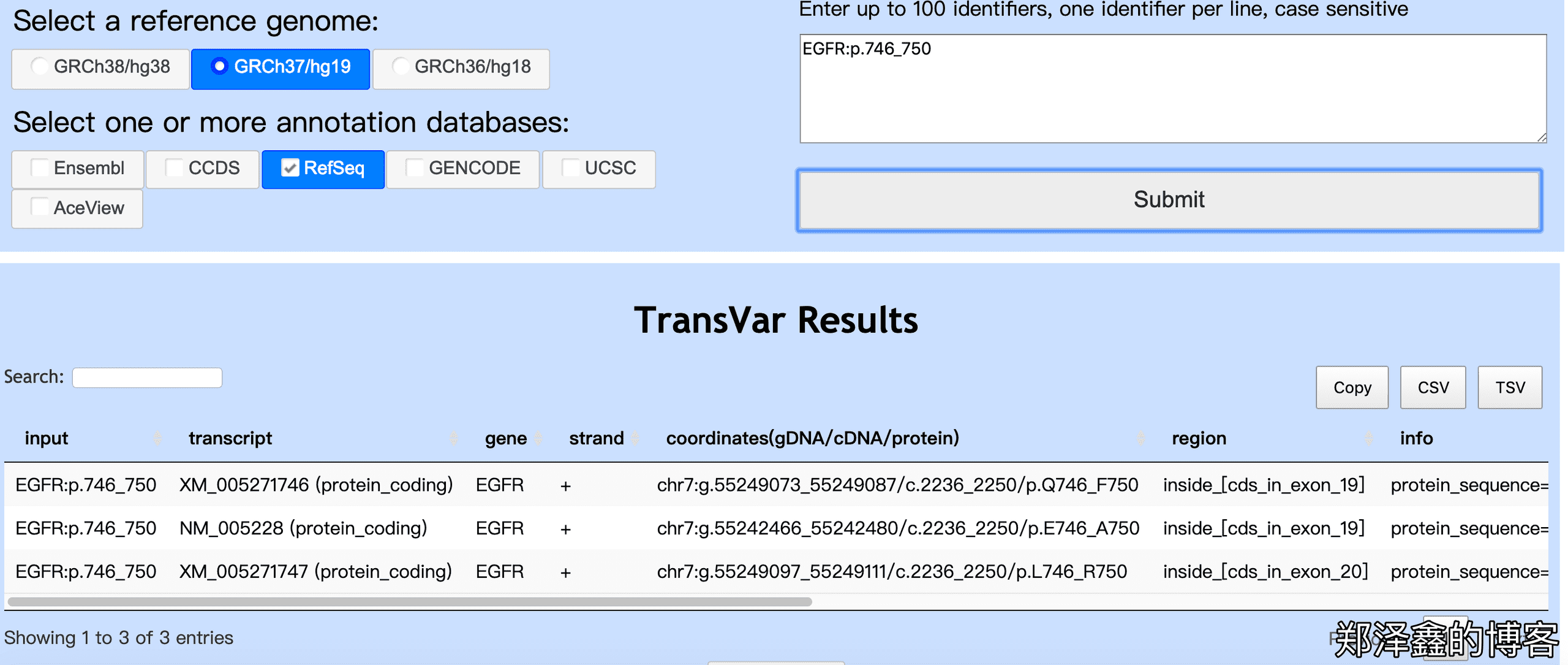
另外,当我们输入为EGFR:p.746_750时,如下图我们看到 Transvar 依然可以给我们转换出这个密码子的基因组水平的坐标范围以及 cDNA 水平的坐标范围,这在我们需要通过密码子来查找对应的基因组范围时特别有用。
终端版使用
这么有用的工具,即使在网页上上传输入文件批量转换坐标,对于我们生信工作者来说,有时仍然有些麻烦。
那么能否将 Transvar 部署到本地呢?答案是肯定的!
Transvar 提供了本地部署的方法:
旧版的代码托管主页放在了 SourceTree,https://bitbucket.org/wanding/transvar,last modified 2016-04-12
而新版的代码托管则迁移到了 Github 上,https://github.com/zwdzwd/transvar,并且用户文档位于Read the Docs。
而且还提供了 Docker 镜像。
安装
参考用户文档,安装 Transvar 主要分pip安装和Docker 镜像2种;但安装后需要下载配置 reference 数据库。
使用Python pip进行安装:
sudo pip install transvar # 全局安装
pip install --user transvar # 用户安装使用 Docker 镜像进行安装
docker pull zhouwanding/transvar:2.4.6
docker run -v ~/references/hg38:/data -ti zhouwanding/transvar:2.4.6 transvar panno -i PIK3CA:p.E545K --ensembl --reference /data/hg38.fa
# -v 加载~/references/hg38到 Docker Container 中的/data路径
# transvar panno -i PIK3CA:p.E545K --ensembl --reference /data/hg38.fa 为注释调用命令下载配置 Reference 数据库也很简单,以hg19为例
# set up databases
transvar config --download_anno --refversion hg19
# in case you don't have a reference
transvar config --download_ref --refversion hg19
# in case you do have a reference to link
transvar config -k reference -v [path_to_hg19.fa] --refversion hg19也可以自己自定义配置数据库,具体设置请参考Setup and Customize。
另外,由于直接使用Transvar的命令下载参考数据库有点像ANNOVAR,容易因网络问题出错,因此你也可以到http://transvar.info/transvar_user/annotations/直接自行下载,然后配置(PS:默认的hg19的 dbSNP 数据库是2016年的,而2018年dbSNP v150等SNP 数量直接翻一番,所以建议自行重新下载)。
使用
使用命令也很清晰明了,由于与网页版类似,所以只列出对应的命令,更详细的内容请参考用户文档。
transvar ganno --ccds -i 'chr3:g.178936091G>A' # 基因组正向注释
transvar canno --ccds -i 'PIK3CA:c.1633G>A' # cDNA反向注释
transvar panno -i 'PIK3CA:p.E545K' --ensembl # 氨基酸反向注释
# 其中--ccds、--ensembl为使用不同的数据库,如网页版,可以同时多选,\
# 如 --ccds --ensembl --refseq --ucsc 来进行多选
# 批量注释
## 样式一
# cat data/small_batch_input
# chr3:g.178936091G>A
transvar ganno -l data/small_batch_input --ccds # 直接-l输入格式已转换好的输入文件
## 样式二
# cat data/small_batch_input
# chr3 178936091 G A CCDS43171
# chr9 135782704 C G CCDS6956
transvar ganno -l data/small_batch_input -g 1 -n 2 -r 3 -a 4 -t 5 --ccds
# 指定-g、-n、-r、-a、-t对应的列(染色体、位置、ref、alt、transcript)
## 样式三
# cat data/small_batch_hgvs
# CCDS43171 chr3:g.178936091G>A
# CCDS6956 chr9:g.135782704C>G
transvar ganno -l data/small_batch_hgvs -m 2 -t 1 --ccds
# 类似样式一,-t可选,指定-t、-m对应的列(mutation、transcript)总结
以上,我们可以看到 Transvar 作为坐标转换的神器的作用,特别是大批量的基因组区域、突变需要进行转换/注释时,甚至能指定 VCF 格式的输出。Transvar 其实16年甚至更早就已经发布,一开始源码放在Bitbucket上,经历了很长一段时间的没有更新,后来又转到 Github 上,令人可喜地发现最近又频繁更新起来。
