记录 VEP 关于 COSMIC 注释的一个坑
工作中有用到VEP,确实是一款出色的注释软件。相比ANNOVAR,最大的优点是支持HGVS标准的注释。
可能由于ANNOVAR面世比较早,当时很多变异描述的标准还没有通行,ANNOVAR采取的是按照基因组来left-normalization的方法,而HGVS的标准,则是根据基因/转录本的方向来left-normalization,因此有可能有的基因与ANNOVAR注释的方向相反。在某些地方,如delins的突变,ANNOVAR与VEP的注释也会不相同,当然,我们还是认为两者都是很出色的注释软件。(关于注释软件的比较,可以看看Golden Helix的这篇文章,http://blog.goldenhelix.com/goldenadmin/the-sate-of-variant-annotation-a-comparison-of-annovar-snpeff-and-vep/)
这次,主要是记录一个VEP关于COSMIC注释的坑,避免大家以后踩到。事情的缘由,还是Ryu的一个同事,发现在一个Somatic突变上,VEP注释了多个COSMIC记录,而查证后却发现,很多事不正确的。
比如,我们用编号为COSM476的BRAF:V600E来举例:
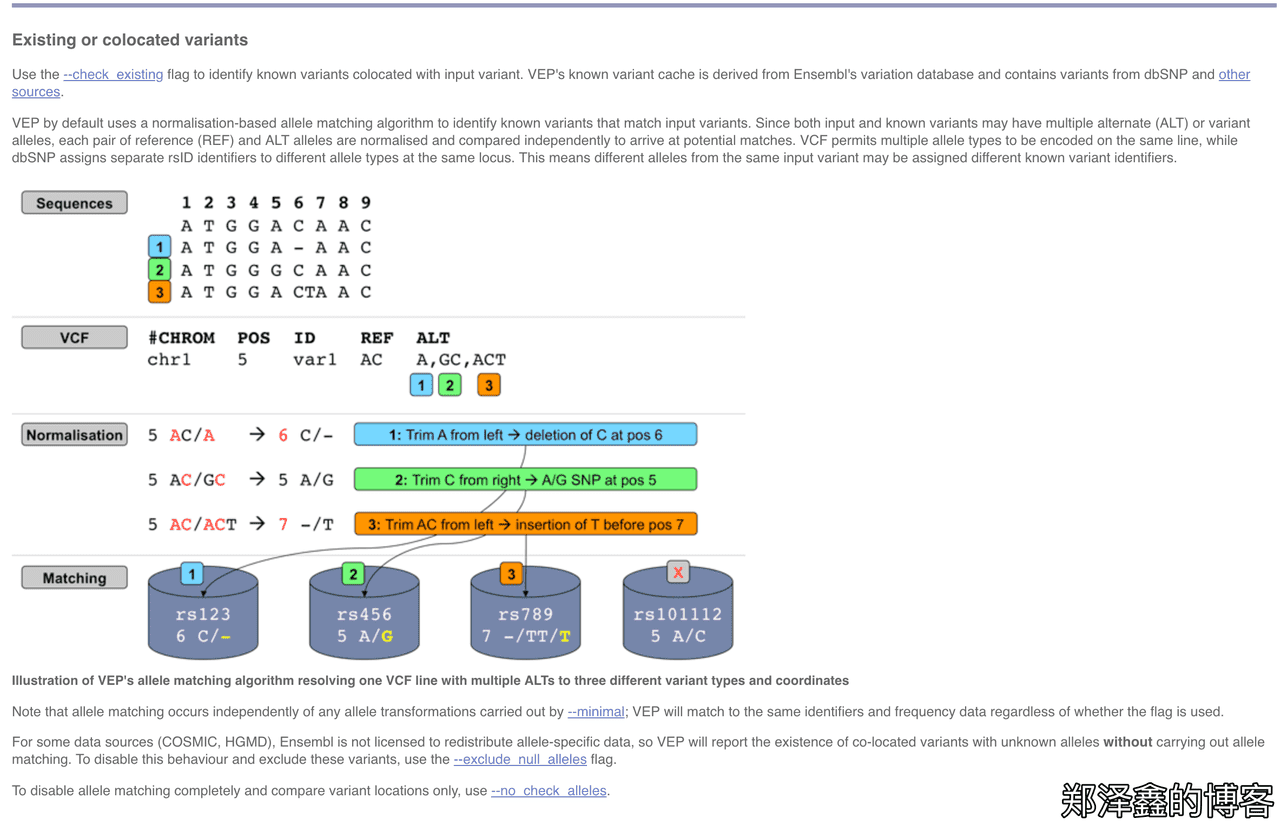
可见,VEP确实注释了多个COSMIC ID,但只有COSM476才是正确的。What?这是怎么发生的。
于是Ryu就去查VEP的文档,终于被我发现了原因。VEP注释是可以注释COSMIC的记录的,但是这个注释,主要是根据染色体坐标而进行的co-located variants注释,没有判断这个突变是什么碱基变化的。

那么为什么要这么做呢?VEP明显是可以实现分辨碱基突变的功能的。援引VEP的原文,原来Ensembl并没有COSMIC、HGMD等数据库的精细到碱基突变的使用许可协议,因此这些数据能够被注释,但是却是换了一个方法,只能精细到染色体位置的层级。
For some data sources (COSMIC, HGMD), Ensembl is not licensed to redistribute allele-specific data, so VEP will report the existence of co-located variants with unknown alleles without carrying out allele matching. To disable this behaviour and exclude these variants, use the --exclude_null_alleles flag.
看到这里,不禁感叹外国人对于版权的意识。在国内,可能COSMIC的注释,分分钟就被人注释上去了,也不会有人追究。其实ANNOVAR的COSMIC注释也止步更新于V70,而现在的COSMIC版本都已经快V90了。
大家以后使用VEP的时候,不要直接使用注释其中的COSMIC记录哦。
