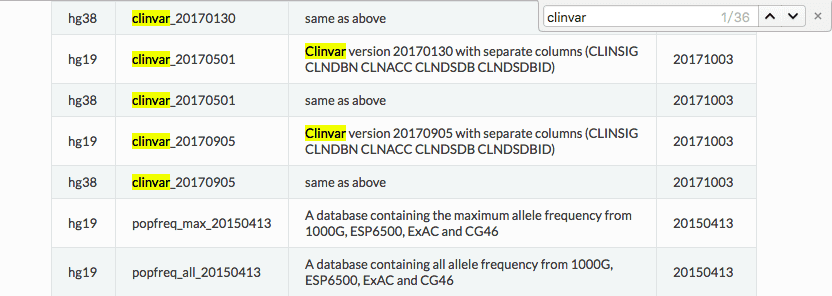
ANNOVAR (2): 关于注释数据库
使用 ANNOVAR 来注释基因组中的突变,首先要下载 ANNOVAR 的注释数据库。如果你需要经常地更新注释数据库,又或者你是“不更新会死星人”,想使用最新的数据库,那么这篇文章也许对你有帮助。
Link
PyInstaller 打包 python 脚本的一些心得
因为在公司经常要帮同事做一个从excel表格中提取出需要的内容的重复工作,比较繁琐还容易出错;于是就想着要写个程序,但是同事又不可能在电脑上也装上python以及相关的包依赖(别人一看就觉得太麻烦而且太冗余),于是就想着将写好的python脚本打包成exe,直接双击使用,方便快捷。
Link

伊恩结
受『[比特新声](http://www.bitvoice.xyz/)』安利,平日里会时不时地看一下『[利器](http://liqi.io/)』,这是『离线』杂志搞的一个网站,一开始是采访优秀的创造者,邀请他们来分享工作时所使用的工具,以及使用工具的方式和原则;然后慢慢丰富到有周报,群分享等内容,可以说是越来越丰富
Linkannovar 注释软件
ANNOVAR是由王凯编写的一个注释软件,可以对SNP和indel进行注释,也可以进行变异的过滤筛选。ANNOVAR能够利用最新的数据来分析各种基因组中的遗传变异。主要包含三种不同的注释方法,Gene-based Annotation(基于基因的注释)、Region-based Annotation(基于区域的注释)、Filter-based Annotation(基于筛选的注释)。
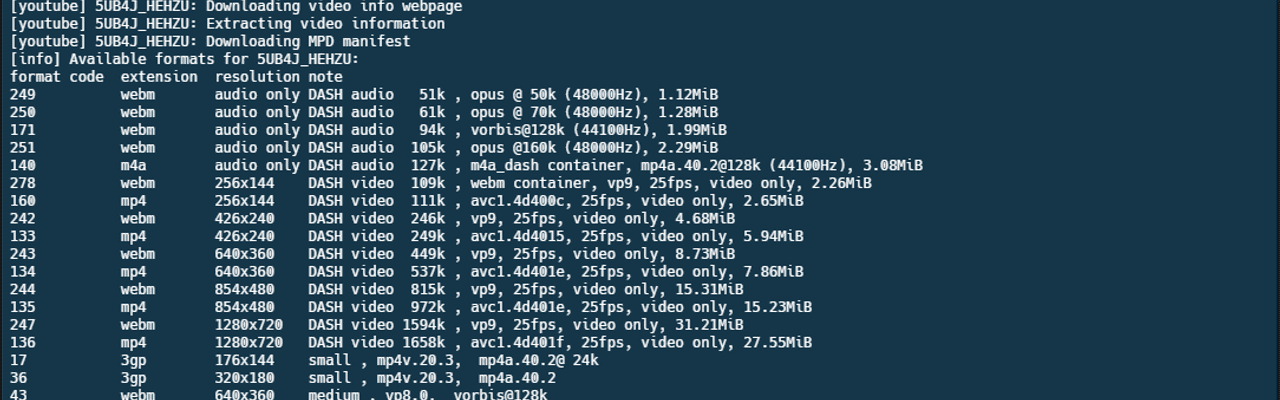
LinkPileup Format 学习笔记
Pileup format is first used by Tony Cox and Zemin Ning at the Sanger Institute. It desribes the base-pair information at each chromosomal position. This format facilitates SNP/indel calling and brief alignment viewing by eyes.
Link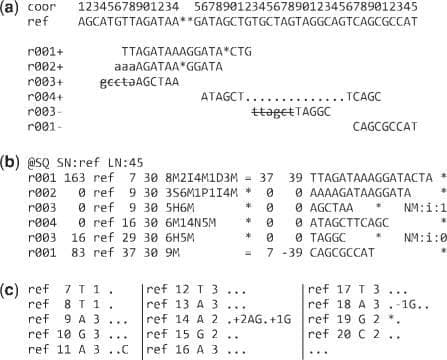
SAM 文件学习笔记
SAM stands for Sequence Alignment/Map format. It is a TAB-delimited text format consisting of a header section, which is optional, and an alignment section. If present, the header must be prior to the alignments. Header lines start with ‘@’, while alignment lines do not. Each alignment line has 11 mandatory fields for essential alignment information such as mapping position, and variable number of optional fields for flexible or aligner specific information.
Link